机器学习——K-Means算法优化(一)代价函数

机器学习——K-Means算法优化(一)代价函数
文章目录
- 机器学习——K-Means算法优化(一)代价函数
-
- 一、K-Means算法(代价函数)
- 二、代码部分
在K-Means算法中,对K个质心的选择,容易陷入局部最小值,从而每次聚类得到不同的结果。
一、K-Means算法(代价函数)
使用多次的随机初始化,并计算每一次建模得到的代价函数值,选取最小的代价函数值作为聚类结果,代价函数公式如下
J ( c ( 1 ) , … , c ( m ) , μ 1 , … , μ K ) = 1 m ∑ i = 1 m ∣ ∣ x ( i ) − μ c ( i ) ∣ ∣ 2 J(c^{(1)},\\dots,c^{(m)},\\mu_{1},\\dots,\\mu_{K})=\\frac{1}{m}\\sum^m_{i=1} {||x^{(i)}-\\mu_c^{(i)}|| } ^2 J(c(1),…,c(m),μ1,…,μK)=m1i=1∑m∣∣x(i)−μc(i)∣∣2
现有数据如下
| 1.658985 | 4.285136 |
|---|---|
| -3.453687 | 3.424321 |
| 4.838138 | -1.151539 |
| … | … |
根据观察,这种数据可以分为4类,即直角坐标系的四个象限。
代码如下:首先还是写一个欧氏距离并初始化质心,最后定义一个kmeans函数。
详情请看之前的文章:
二、代码部分
# 计算距离
def euclDistance(vector1, vector2): return np.sqrt(sum((vector2 - vector1)2))# 初始化质心
def initCentroids(data, k): numSamples, dim = data.shape# k个质心,列数跟样本的列数一样centroids = np.zeros((k, dim)) # 随机选出k个质心for i in range(k): # 随机选取一个样本的索引index = int(np.random.uniform(0, numSamples)) # 作为初始化的质心centroids[i, :] = data[index, :] return centroids # 传入数据集和k的值
def kmeans(data, k): # 计算样本个数numSamples = data.shape[0] # 样本的属性,第一列保存该样本属于哪个簇,第二列保存该样本跟它所属簇的误差clusterData = np.array(np.zeros((numSamples, 2))) # 决定质心是否要改变的变量clusterChanged = True # 初始化质心 centroids = initCentroids(data, k) while clusterChanged: clusterChanged = False # 循环每一个样本 for i in range(numSamples): # 最小距离minDist = 100000.0 # 定义样本所属的簇minIndex = 0 # 循环计算每一个质心与该样本的距离for j in range(k): # 循环每一个质心和样本,计算距离distance = euclDistance(centroids[j, :], data[i, :]) # 如果计算的距离小于最小距离,则更新最小距离if distance < minDist: minDist = distance # 更新样本所属的簇minIndex = j # 更新最小距离clusterData[i, 1] = distance# 如果样本的所属的簇发生了变化if clusterData[i, 0] != minIndex: # 质心要重新计算clusterChanged = True# 更新样本的簇clusterData[i, 0] = minIndex# 更新质心for j in range(k): # 获取第j个簇所有的样本所在的索引cluster_index = np.nonzero(clusterData[:, 0] == j)# 第j个簇所有的样本点pointsInCluster = data[cluster_index] # 计算质心centroids[j, :] = np.mean(pointsInCluster, axis = 0)
# showCluster(data, k, centroids, clusterData)return centroids, clusterData # 显示结果
def showCluster(data, k, centroids, clusterData): numSamples, dim = data.shape if dim != 2: print("dimension of your data is not 2!") return 1 # 用不同颜色形状来表示各个类别mark = ['or', 'ob', 'og', 'ok', '^r', '+r', 'sr', 'dr', '<r', 'pr'] if k > len(mark): print("Your k is too large!") return 1 # 画样本点 for i in range(numSamples): markIndex = int(clusterData[i, 0]) plt.plot(data[i, 0], data[i, 1], mark[markIndex]) # 用不同颜色形状来表示各个类别mark = ['*r', '*b', '*g', '*k', '^b', '+b', 'sb', 'db', '<b', 'pb'] # 画质心点 for i in range(k): plt.plot(centroids[i, 0], centroids[i, 1], mark[i], markersize = 20) plt.show()
随后设置K的值为4,并写一个上述的代价函数来对样本点进行迭代。
# 设置k值
k = 4 min_loss = 10000
min_loss_centroids = np.array([])
min_loss_clusterData = np.array([])for i in range(50):# centroids 簇的中心点 centroids, clusterData = kmeans(data, k) loss = sum(clusterData[:,1])/data.shape[0]if loss < min_loss:min_loss = lossmin_loss_centroids = centroidsmin_loss_clusterData = clusterDatacentroids = min_loss_centroids
clusterData = min_loss_clusterData# 显示结果
showCluster(data, k, centroids, clusterData)
结果如下:
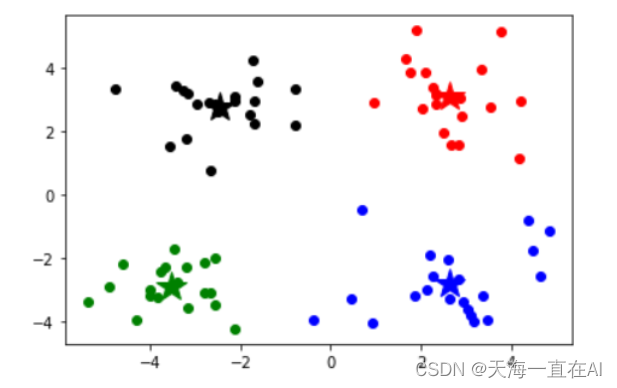
随后我们将每个簇的作用域标记出来,先写出预测函数
# 做预测
x_test = [0,1]
np.tile(x_test,(k,1))
# 误差
np.tile(x_test,(k,1))-centroids
# 误差平方
(np.tile(x_test,(k,1))-centroids)2
# 误差平方和
((np.tile(x_test,(k,1))-centroids)2).sum(axis=1)
# 最小值所在的索引号
np.argmin(((np.tile(x_test,(k,1))-centroids)2).sum(axis=1))
def predict(datas):return np.array([np.argmin(((np.tile(data,(k,1))-centroids)2).sum(axis=1)) for data in datas])
画出簇的作用域
# 获取数据值所在的范围
x_min, x_max = data[:, 0].min() - 1, data[:, 0].max() + 1
y_min, y_max = data[:, 1].min() - 1, data[:, 1].max() + 1# 生成网格矩阵
xx, yy = np.meshgrid(np.arange(x_min, x_max, 0.02),np.arange(y_min, y_max, 0.02))z = predict(np.c_[xx.ravel(), yy.ravel()])# ravel与flatten类似,多维数据转一维。flatten不会改变原始数据,ravel会改变原始数据
z = z.reshape(xx.shape)
# 等高线图
cs = plt.contourf(xx, yy, z)
# 显示结果
showCluster(data, k, centroids, clusterData)
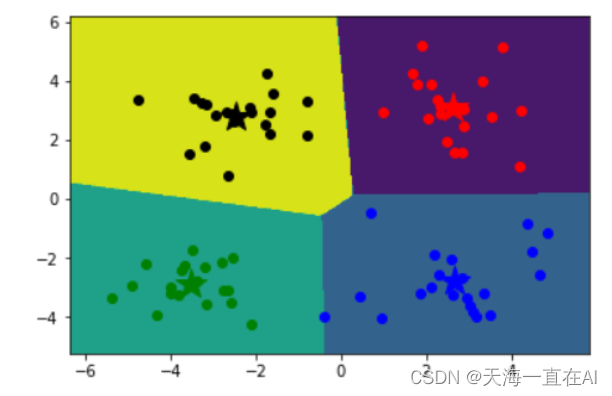
最后分成了类似坐标系的四块区域。
