arxiv2017 | 用于分子神经网络建模的数据增强 SMILES Enumeration


论文标题:SMILES Enumeration as Data Augmentation for Neural Network Modeling of Molecules
论文地址:https://arxiv.org/abs/1703.07076
代码地址:https://github.com/Ebjerrum/SMILES-enumeration
一、摘要

摘要中明显提出:先指出多个SMILES对应一个分子,标准SMILES对应一个分子。采用数据增强,使得数据集扩大130倍,然后指出改进效果。
二、Introduction
第一段:描述数据集大小限制了OSAR领域的应用。小数据集需要更多正则化或者小网络。在CV中可以通过多种手段进行数据增强,以扩大数据集,使得模型更具鲁棒性。
第二段:分子表征有三种(分子描述符、SMILES、Graph),SMILES的变化有很多种,如CCC→C(C)C,分子越复杂,其变化越多,对应的SMILES也越多。如下:
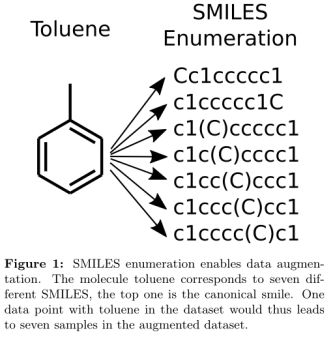
第三段:描述应用方法。SMILES enumeration for QSAR using LSTM
三、Methods
SMILES enumeration:一个python脚本(函数)。将SMILES转化为molfile→打乱原子顺序→转换为mol→RDKit生成SMILES→存入set中(保证不重)。简洁代码:
def randomize_smile(sml):"""Function that randomizes a SMILES sequnce. This was adapted from theimplemetation of E. Bjerrum 2017, SMILES Enumeration as Data Augmentationfor Neural Network Modeling of Molecules.Args:sml: SMILES sequnce to randomize.Return:randomized SMILES sequnce ornan if SMILES is not interpretable."""try:m = Chem.MolFromSmiles(sml)ans = list(range(m.GetNumAtoms()))np.random.shuffle(ans)nm = Chem.RenumberAtoms(m, ans)return Chem.MolToSmiles(nm, canonical=False)except:return float('nan')
Molecular dataset:756 dihydrofolate inhibitors with P. carinii DHFR inhibition data
J. J. Sutherland, L. A. O’Brien, D. F. Weaver, Spline-fitting with a genetic algorithm: a method for developing classification structure-activity relationships., Journal of chemical information and computer sciences 43 (2003) 1906–1915. doi: 10.1021/ci034143r.
训练:测试=9:1,并没有在验证集上取best。embedding_dim = 74,one-hot编码。
LSTM neural network:LSTM+全连接层。两个模型,一个是标准模型,一个是枚举模型。实行超参数搜索。标准模型应该LSTM layers更小会更好一点(因为数据集小),应该是陷入模型局部最优值。但是L1、L2的正则化有一点的作用。
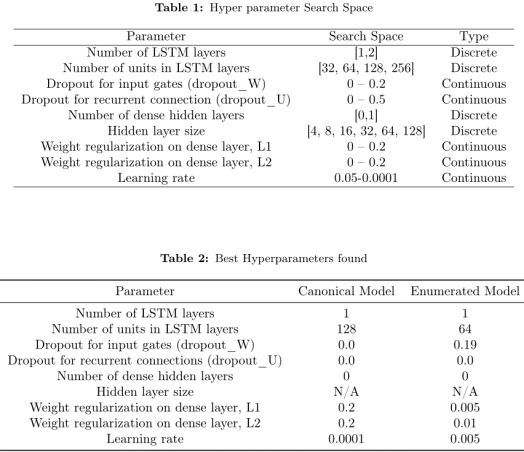
损失下降:蓝线是没有正则化惩罚的均方误差,绿线是包含正则化惩罚的损失,红线是测试集中的均方误差。标准模型在标准数据上要迭代更多epoch,因为数据集要远小于枚举数据集,需要更多梯度更新。但是运行时间大致相同。
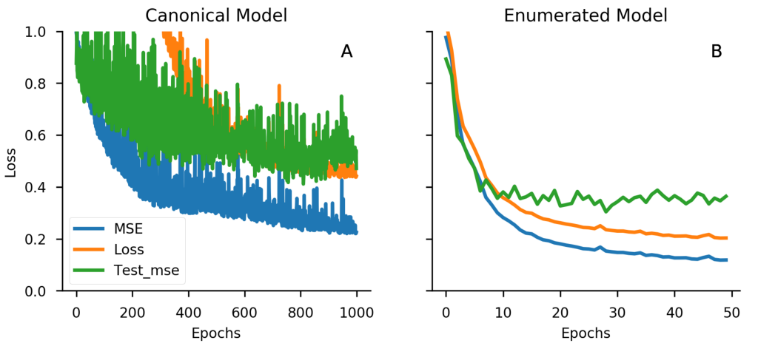
散点图:左列为标准模型在标准数据集、枚举数据集上的表现。右列为枚举模型在标准数据集、枚举数据集上的表现。
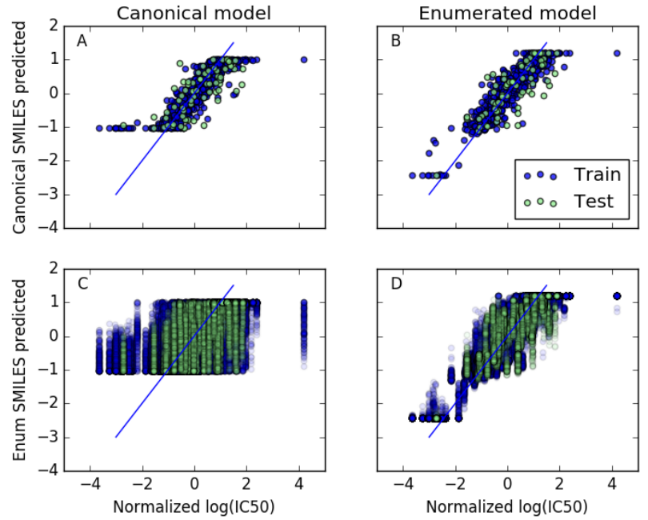
下表列出具体数值:
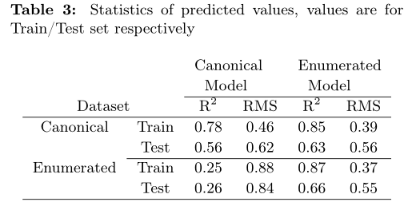
该研究缺乏对训练集、测试集和验证集的划分,其中超参数在测试集上进行调优,但最终性能在验证集上进行评估。因此,LSTM-QSAR模型观测到的预测性能可能在一定程度上被高估。
然而,本研究的重点是使用SMILES枚举的增益,而不是生成最优的DHFR QSAR模型。正则模型在训练和测试集上的性能都较低。如果性能上的差异是由于过度拟合造成的,那么较小的数据集可能会有优势。
四、Conclusion
This short investigation has shown promise in using SMILES enumeration as a data augmentation technique for neural network QSAR models based on SMILES data.
