论文解读:4mCBERT:基于集成学习策略,通过序列和化学衍生信息识别DNA n4 -甲基胞嘧啶位点的计算工具

Title:4mCBERT: A computing tool for the identification of DNA N4-methylcytosine sites by sequence- and chemical-derived information based on ensemble learning strategies
期刊: International Journal of Biological Macromolecules
影响因子:8.025
中科院分区:2区
出版日期: 2023-03-01
Github:https://github. com/abcair/4mCBERT
摘要
N4 -甲基胞嘧啶(N4-methylcytosine, 4mC)。在本文中,我们提出了一个称为4mCBERT的模型,该模型通过序列特征one-hot、EIIP、NCP、word2vec、PCP、chemical BERT编码DNA序列片段,并使用集成学习框架开发了一个预测模型。首先构建了PCP特征和化学BERT特征,并将其应用于4mC位点预测,对识别4mC有积极贡献。对于MCC,4mCBERT在六个独立的基准数据集上显著优于其他最先进的模型,包括A.taliana, C.elegans, D.melanogaster, E.coli, G.Pickering和G.subterraneous。提高4.32%至24.39%,2.52%至31.65%,2%至16.49%,6.63%至35.15%,8.59%至61.85%,8.45%至34.45%。此外,4mCBERT旨在允许用户预测4mC站点并重新训练4mC预测模型。简而言之,4mCBERT通过结合序列和化学驱动信息,在六个基准数据集上显示出更高的性能。
前人的工作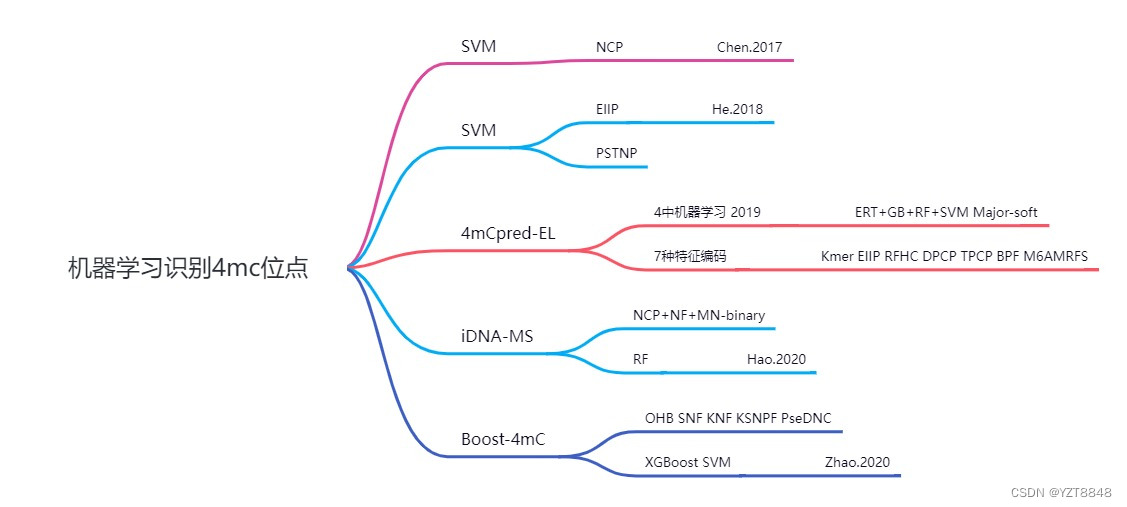
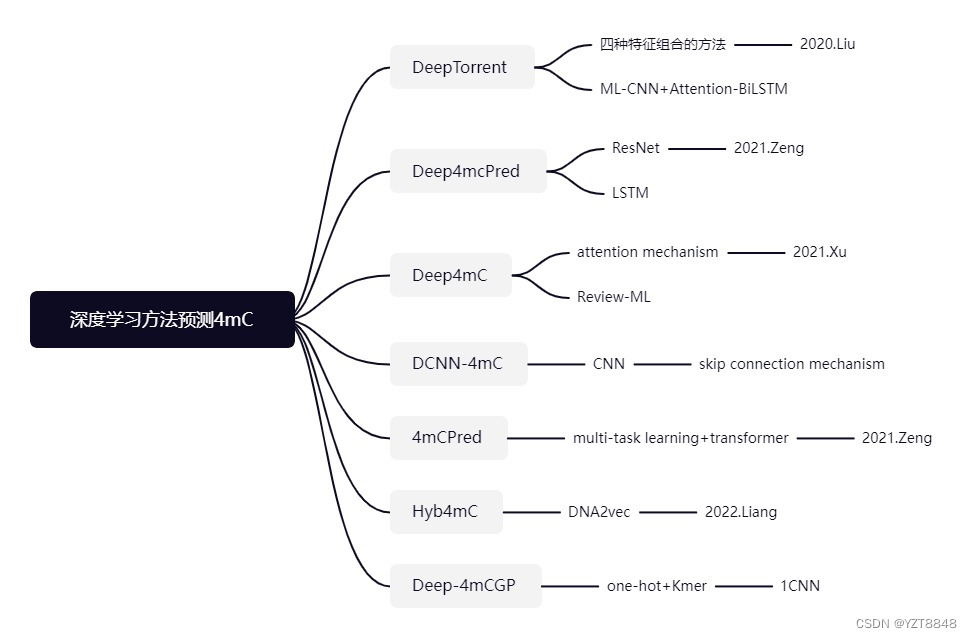
方法和数据集
数据集
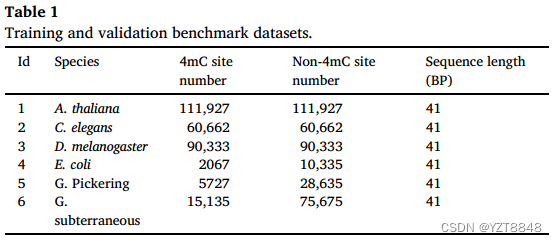
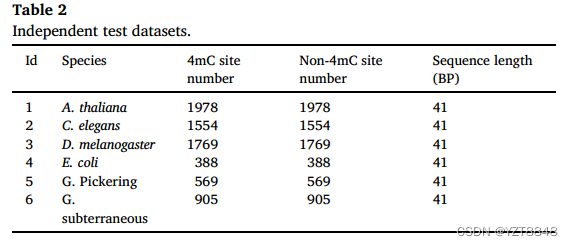
两个基准数据集来自Xu的工作,由MethSMRT数据库构建。第一个基准数据集包含6个物种的4mC位点序列,包括A. thaliana、C. elegans、D. melanogaster、E. coli、G. Pickering和G. subterraneous,如表1所示。将每个物种的第一个基准数据集分成80%用于训练,20%用于验证。第二个基准数据集也来自Xu的工作,作为独立的测试数据集进行公平的调查和比较,其中包含从MethSMRT数据集中新收集的6个物种的4mC位点序列。第二个基准数据集进行修改置信度评分(QV)测试,如果QV评分大于30,则序列将被保留。为了消除相似度冗余,执行CD-HIT对相似度超过70%的序列进行过滤。对于6个物种,基准数据集中的每个序列长度为41个碱基对(bp) DNA片段。两个基准数据集的详细信息分别列在表1和表2中。
方法
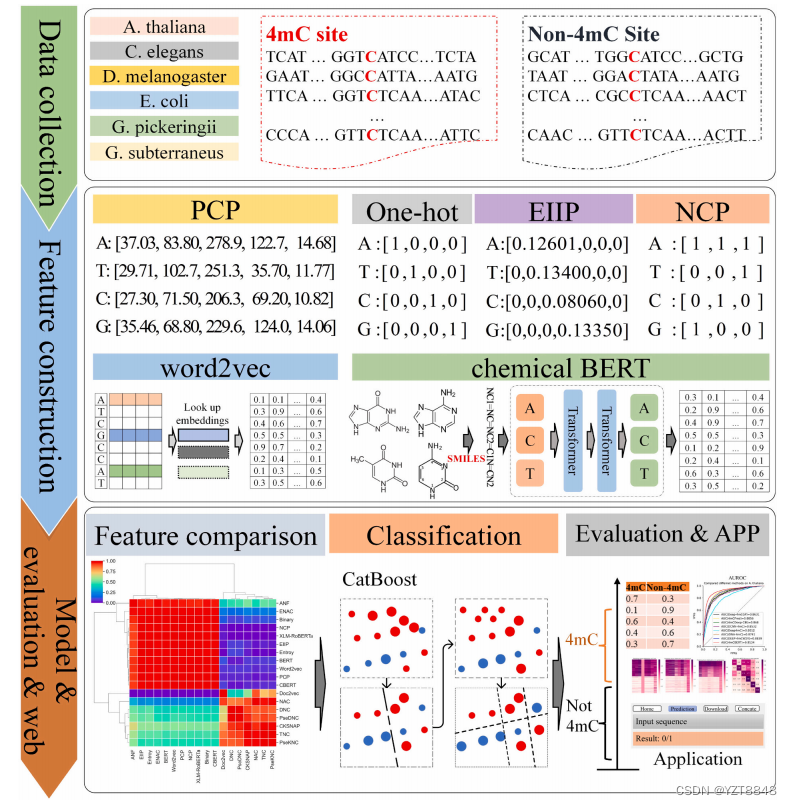
在本文中,我们引入了一种新的预测模型4mCBERT来识别4mC位点。4mCBERT的输入是六种精心手工制作的特征,包括one-hot, EIIP, NCP, word2vec, PCP和化学BERT。训练模型基于梯度增强决策树CatBoost。4mCBERT的总体框架如图3所示,共分为五个步骤。首先收集并处理了A. thaliana、C. elegans、D. melanogaster、E. coli、G. Pickering和G. subterraneous 6个物种的数据集。对于每个物种,都有两个基准数据集,分别称为基准dataset1和基准dataset2。将每个物种的基准数据集1分成80%和20%的训练数据集和验证数据集。
每个物种的基准数据集是一个独立的测试数据集,用于评估4mCBERT的性能。第二步是将DNA序列片段转换为数字载体。One-hot、EIIP、NCP、word2vec和PCP、化学BERT六种埋埋方法经过精心选择和构建。此外,与其他广泛使用的编码方法相比,one-hot、EIIP、NCP和word2vec在预测4mC位点方面表现出更优的效果。分子结构信息启发了PCP和化学BERT的构建。PCP利用分子物理特性编码DNA序列片段,化学BERT利用NLP技术学习分子结构信息。最后,对one-hot (4 × 41 = 164维)、EIIP (4 × 41 = 164维)、NCP (4 × 41 = 164维)、word2vec (4 × 41 = 164维)、PCP (5 × 41 = 205维)和chemical BERT (3 × 16 × 41 = 1968维)进行平化并合并成2788维的直线向量。然后引入CatBoost训练集成学习模型。CatBoost是一种新的梯度增强决策树(GBDT)算法,它可以处理训练阶段的特征,包括构建决策树的两个步骤。第一步是塑造树结构,第二步是为固定的叶子节点赋值。基于贪心策略,CatBoost在为当前树创建分裂点时考虑候选组合以提高精度。此外,CatBoost可以在多线程上运行,以加速大数据集上的训练过程。
结果
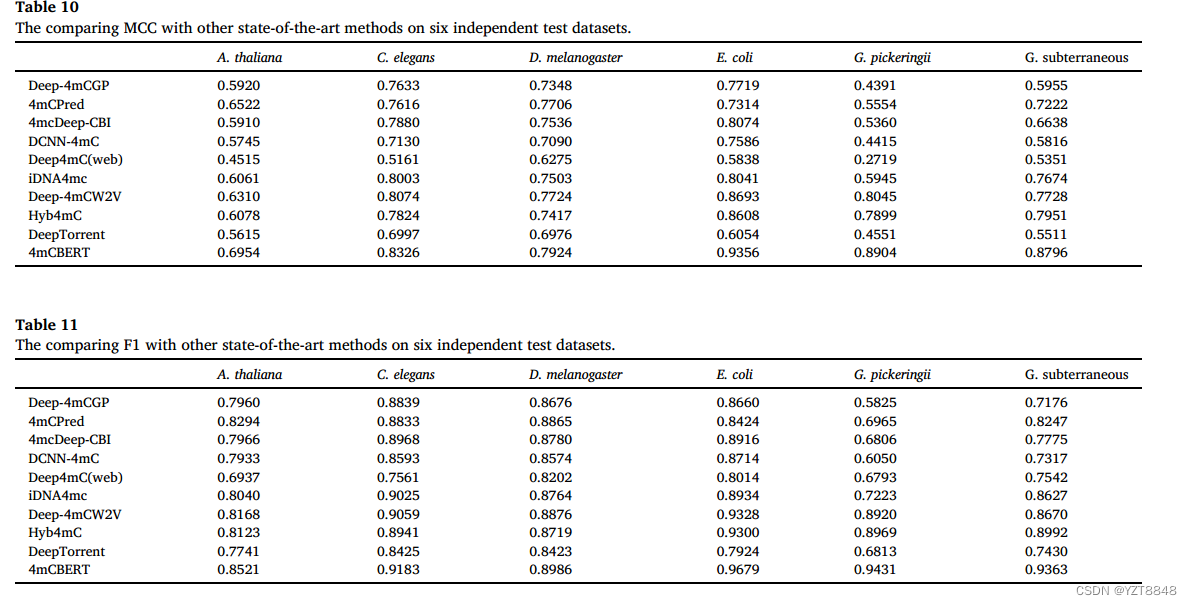
接下来,我们将我们的模型与其他最先进的模型进行了比较,包括Deep- 4mcgp , 4mCPred, 4mcDeep-CBI, DCNN-4mC, Deep4mC, iDNA4mc, Deep- 4mCW2V, Hyb4mC和DeepTorrent[23]在独立的测试数据集上,包括A. thaliana, C. elegans, D. melanogaster, E. coli, G.。皮克林和G.地下六种。为了做一个公平的比较,8对于每个物种,比较模型分别在相同的训练数据集上训练,然后在相同的独立测试数据集上测试。根据预测结果计算各物种模型的评价指标值。表10和表11显示了4mCBERT和其他最新模型在每个独立测试数据集上的MCC和F1分数。
实验过程
特征提取
1、Binary 2、NCP 3、word2vec
PCP
在本文中,我们引入了物理化学性质(PCP)特征来表示DNA序列片段。PCP特征侧重于每个核苷酸的物理化学性质,而不是整个序列。物理化学性质包括摩尔折射率、摩尔体积、等比体积、表面张力和极化率。对于“A”、“T”、“C”和“G”四种核苷酸,其理化性质如表4所示。根据表4,长度为的DNA序列片段可以编码到L× 5矩阵中。值得注意的是,首先采用PCP嵌入方法来识别4mC位点。
Chemical Bert
为了进一步挖掘贡献的特征来区分4mC和非4mC,我们引入了一种流行的方法,称为双向编码器表示变压器(BERT)来挖掘潜在的特征。BERT是谷歌在2018年开发的一种基于变压器的NLP预训练机器学习技术。受现代计算化学的启发,化学分子的数字矢量可以通过计算机科学计算出来。每种化学分子都有其特定的结构和成分。这些特征为DNA片段的新表示提供了机会。例如,苯具有6个碳原子和6个氢原子,呈环状空间结构,用简化分子输入线输入系统(SMILES)格式可简写为“C1 = CC = CC = C1”。对于DNA序列片段,它是由“a”,“T”,“C”和“G”的不同排列和组合构成的。因此,要对DNA序列进行编码,首要任务是将“A”、“T”、“C”和“G”编码为数值向量。如图2所示,“A”和“G”包含两个环结构,“T”和“C”只包含一个环结构,只有“T”包含“CH3”。
这些差异提供了代表每个核苷酸的单独信息。为了保存每个核苷酸的结构信息,使用SIMILES表示化学物种的结构,将多边形信息转换并保存为一行字符串。例如核苷酸“A”的分子结构如图2.1所示,SMILES格式可以表示为“NC1 = NC = NC2 = C1N = CN2”。“C”、“G”、“T”可以用“CC1 = CNC(=O)NC1 =O”、“NC1 = C(NC(=O)N = C1)N”和“O = C1C2NCNC2NC(N)N1”表示。下面的过程是将这个具有结构信息的行字符串转换为数字矢量。由于NLP技术,分子亚结构的SMILES格式可以被视为一个“句子”。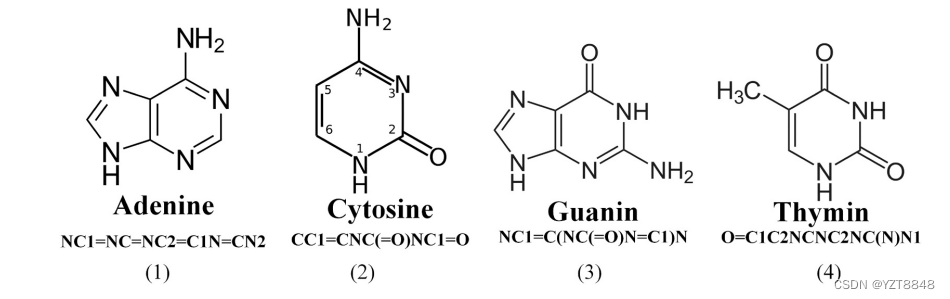
因此,引入化学BERT方法嵌入各个元素来表示分子的子结构信息。选取三种BERT模型分别表示“A”、“T”、“C”和“G”的SMILES。
第一个BERT模型称为ChemBERTa,它是基于RoBERTa转换器的12个注意力头,在PubChem的7700万个独特的SMILES上训练,带有15%的面具。第二个BERT模型称为XLM-RoBERTa,第三个BERT模型称为BERT-base,在一个小型数据集上训练,该数据集包含PubChem提供的150万个独特的SMILES。得到预训练好的BERT模型后,将“A”、“T”、“C”、“G”嵌入到数字向量中。为了避免维度突变,提取前16维向量用于下面的DNA片段表示。据我们所知,分子结构信息首次应用于4mC位点的识别,并可广泛扩展到RNA和蛋白质序列的包埋。
模型
在本文中,我们引入了一种新的预测模型4mCBERT来识别4mC位点。4mCBERT的输入是六种精心手工制作的特征,包括one-hot, EIIP, NCP, word2vec, PCP和化学BERT。训练模型基于梯度增强决策树CatBoost。4mCBERT的总体框架如图3所示,共分为五个步骤。首先收集并处理了A. thaliana、C. elegans、D. melanogaster、E. coli、G. Pickering和G. subterraneous 6个物种的数据集。对于每个物种,都有两个基准数据集,分别称为基准dataset1和基准dataset2。将每个物种的基准数据集1分成80%和20%的训练数据集和验证数据集。
每个物种的基准数据集是一个独立的测试数据集,用于评估4mCBERT的性能。第二步是将DNA序列片段转换为数字载体。One-hot、EIIP、NCP、word2vec和PCP、化学BERT六种埋埋方法经过精心选择和构建。此外,与其他广泛使用的编码方法相比,one-hot、EIIP、NCP和word2vec在预测4mC位点方面表现出更优的效果。分子结构信息启发了PCP和化学BERT的构建。PCP利用分子物理特性编码DNA序列片段,化学BERT利用NLP技术学习分子结构信息。最后,对one-hot (4 × 41 = 164维)、EIIP (4 × 41 = 164维)、NCP (4 × 41 = 164维)、word2vec (4 × 41 = 164维)、PCP (5 × 41 = 205维)和chemical BERT (3 × 16 × 41 = 1968维)进行平化并合并成2788维的直线向量。然后引入CatBoost训练集成学习模型。CatBoost是一种新的梯度增强决策树(GBDT)算法,它可以处理训练阶段的特征,包括构建决策树的两个步骤。第一步是塑造树结构,第二步是为固定的叶子节点赋值。基于贪心策略,CatBoost在为当前树创建分裂点时考虑候选组合以提高精度。此外,CatBoost可以在多线程上运行,以加速大数据集上的训练过程。
受益于上述进展,选择CatBoost模型训练4mC位点识别的预测模型。最后,4mCBERT通过多种指标进行评估,并为学术研究开发了一个用户友好的图形用户界面(GUI)实现和4mCBERT的web服务器。4mCBERT的工作流程如图3所示。
单个特征编码比较
在这部分,我们总结了现有的编码特征,并将其与我们新构建的编码特征(PCP和化学BERT)进行了比较。然后,根据比较结果,我们列出了4mC预测的推荐特征。编码DNA序列片段的常规特征包括单热、核苷酸化学性质(NCP)、增强核酸组成(ENAC)、EIIP、三核苷酸组成(TNC)、k-间隔氨基酸对组成(CKSNAP)[54]、二核苷酸组成(DNC)、伪二核苷酸组成(PseDNC)、伪k-元核苷酸组成(PseKNC)[56]、核酸组成(NAC)、累积核苷酸频率(ANF)[28]、word2vec、Doc2vec,熵。其中one-hot、EIIP、NCP、word2vec、PCP、chemical BERT在2.2部分介绍,其他特性如表S2所示。表5中列出了六个物种数据集上各编码特征的比较(粗体表示top8性能),完整的比较细节列在表S3到表S5中。其中EIIP、NCP、word2vec、PCP和化学BERT特征具有较高的MCC和AUC。
为了选择编码DNA片段的最终特征,我们在六个物种数据集上绘制了这些特征的聚类热图。如图5所示,在不同物种数据集上,one-hot、EIIP、NCP、word2vec、PCP和化学BERT特征聚为一组。因此,基于更突出的性能和亲和力,我们选择one-hot、EIIP、NCP、Word2vec、PCP和化学BERT特征对DNA序列片段进行编码,以识别4mC位点。
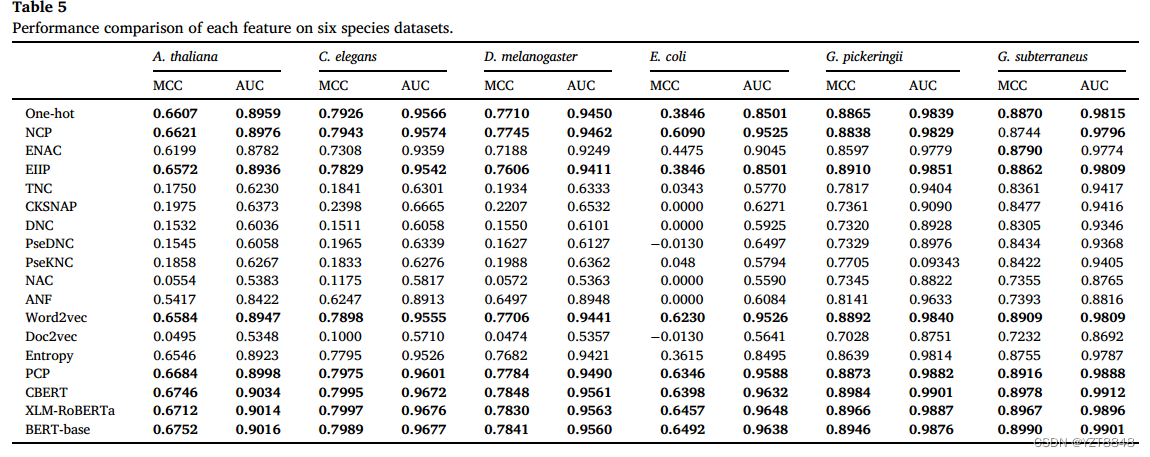
特征融合比较
为了说明PCP和化学BERT的积极贡献,我们比较了one-hot+EIIP+NCP + word2vec、one-hot+EIIP+NCP + word2vec + PCP、one-hot+EIIP+NCP + word2vec +cherimal-BERT和one-hot+EIIP+NCP + word2vec + PCP +cherimal-BERT组合的性能。引入MCC和F1来衡量性能,如表6和表7所示。从表6和表7中我们可以发现,PCP和cherimal-BERT对识别4mC和非4mC确实有积极的贡献,one-hot+EIIP+NCP + word2vec + PCP +cherimal-BERT组合的MCC和F1得分更高。
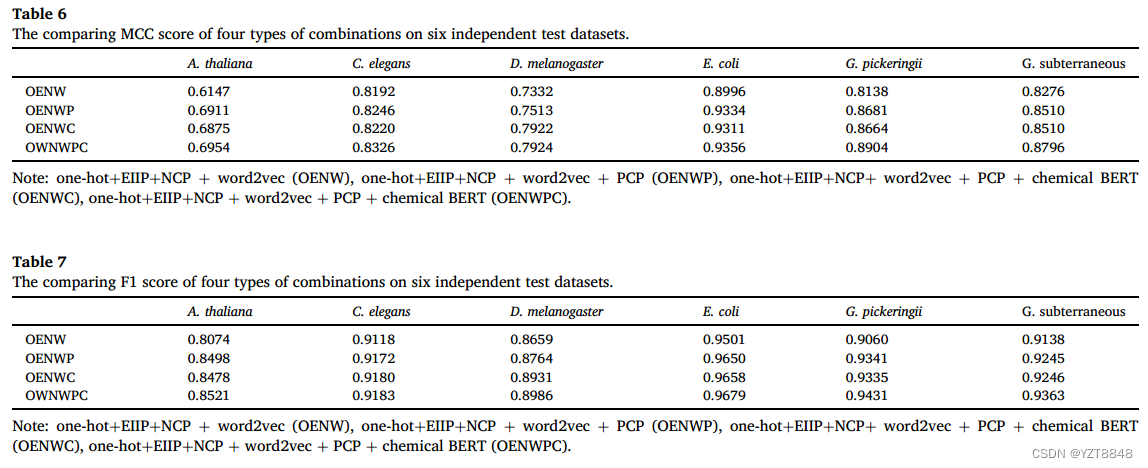
与传统机器学习的比较
对DNA序列段进行特征工程后。采用CatBoost[作为最终预测模型构建4mCBERT。为了评估4mCBERT的预测能力,我们分别在6个物种上引入6个独立的测试数据集来测试4mCBERT。首先,我们将4mCBERT模型与ResNet101 (101 residual blocks)、CNN1D(一维神经网络)、FCN(全连接网络)、SVM(支持向量机)、RF(随机森林)、AdaBoost(自适应增强)等生物学领域广泛应用的主流模型进行比较。为了公平起见,比较方法中所有的训练、验证和测试数据集都与4mCBERT相同,所有比较方法的输入编码特征也与4mCBERT相同。这些比较方法的详细参数如表S6所示。比较MCC和F1列于表8和表9,每种比较方法的详细参数列于表S6。从表8和表9中我们可以发现,以CatBoost为分类器的4mCBERT在六个独立的测试数据集上获得了更高的MCC和F1。RF MCC第二高,F1低于4mCBERT(2.35% ~ 12%)。ResNet101在计算机视觉方面表现出强大的能力,但在4mC预测方面表现不佳。CNN1D也没有得到满意的结果。FCN和SVM表现出相似的结果,但远不如4mCBERT。AdaBoost也是一种提高方法,但在预测4mC时,从MCC和F1的角度来看,表现较差。其他比较包括TN, TP, FP, FN, Precise, SE, SP, ACC和AUC列在表S7至表S11中。
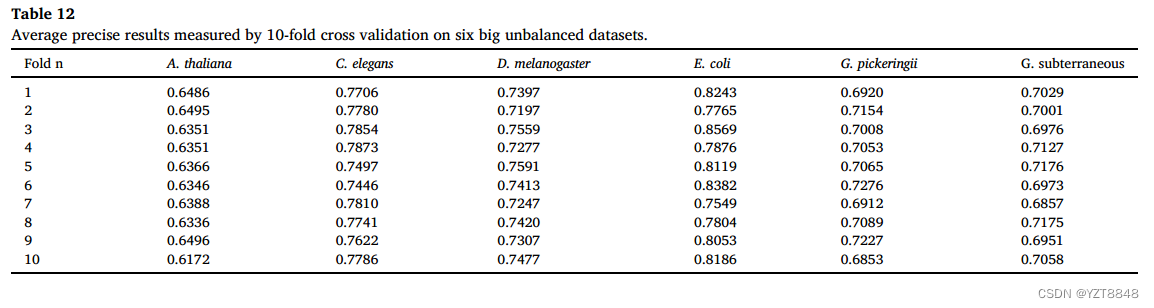
为了详细说明两者的区别,AUROC如图6所示。从图6中我们可以发现4mCBERT的ROC比其他方法更陡,并将其完全包裹起来,这意味着4mCBERT在预测4mC位点时比其他比较方法灵敏度更高。在6个物种数据集中,4mCBERT的AUC评分比其他方法分别高出2.37% ~ 11.85%、1.82% ~ 20.85%、1.79% ~ 7.99%、1.85% ~ 11.51%、2.72% ~ 19.08%和2.9 % ~ 21.66%。通过AUROC对比,我们可以发现基于CatBoost的4mCBERT在预测4mC段时准确率更高。
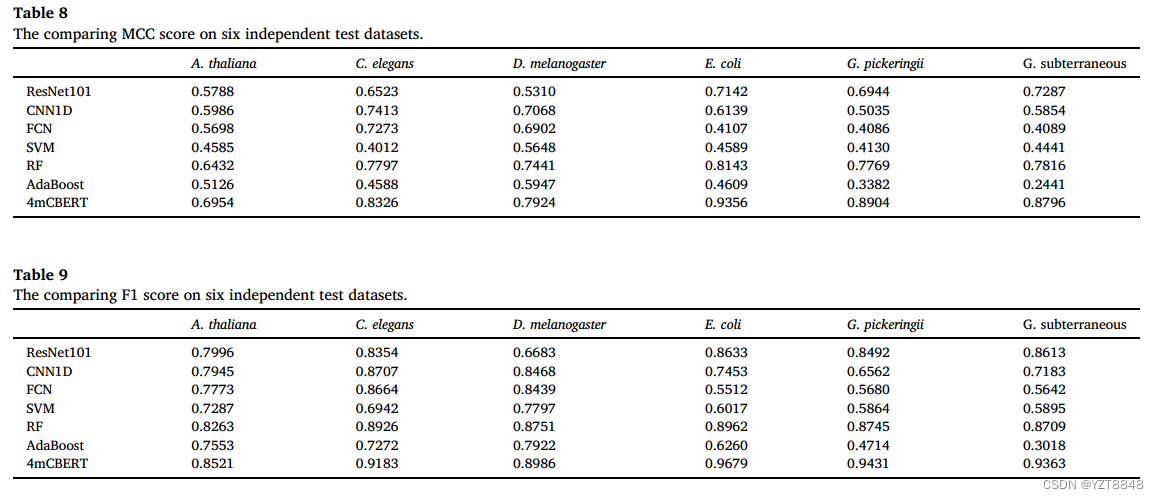
不平衡数据集实验
为了显示4mCBERT在不平衡数据集上的性能,我们通过10倍交叉验证来测量表1数据库上的4mCBERT, 4mC和非4mC的不平衡比例为1:5。在表1所示的大数据集中,a . thaliana、C. elegans和D. melanogaster的数据集是平衡的,E. coli、G. Pickering和G. subterraneous数据集是不平衡的,4mC和非4mC的数量比为1:5。为了构建非平衡数据集,我们对A. thaliana、C. elegans和D. melanogaster的数据集采样1/5 4mC序列来生成非平衡数据集。然后我们利用10倍交叉验证来衡量4mCBERT在这些不平衡数据集上的性能。我们使用平均精度(AP)来衡量4mCBERT在这些不平衡数据集上的性能。结果如表12所示。此外,绘制每条折叠的精度-召回率和ROC曲线(proroc)来显示4mCBERT的性能,如图所示
ROC曲线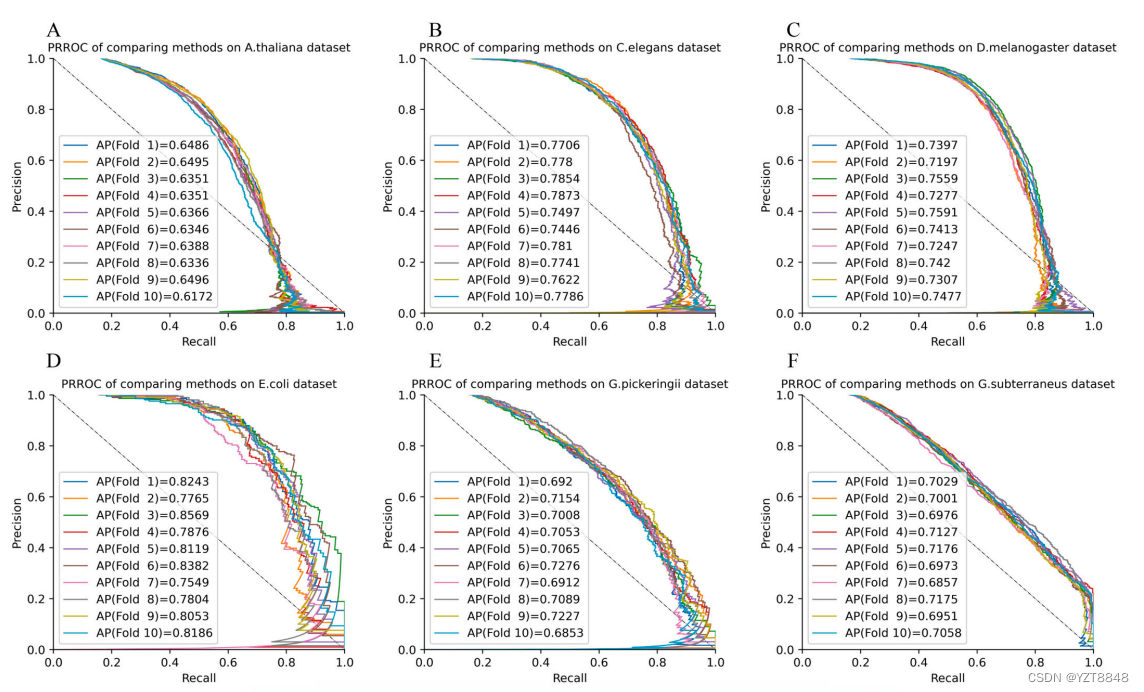
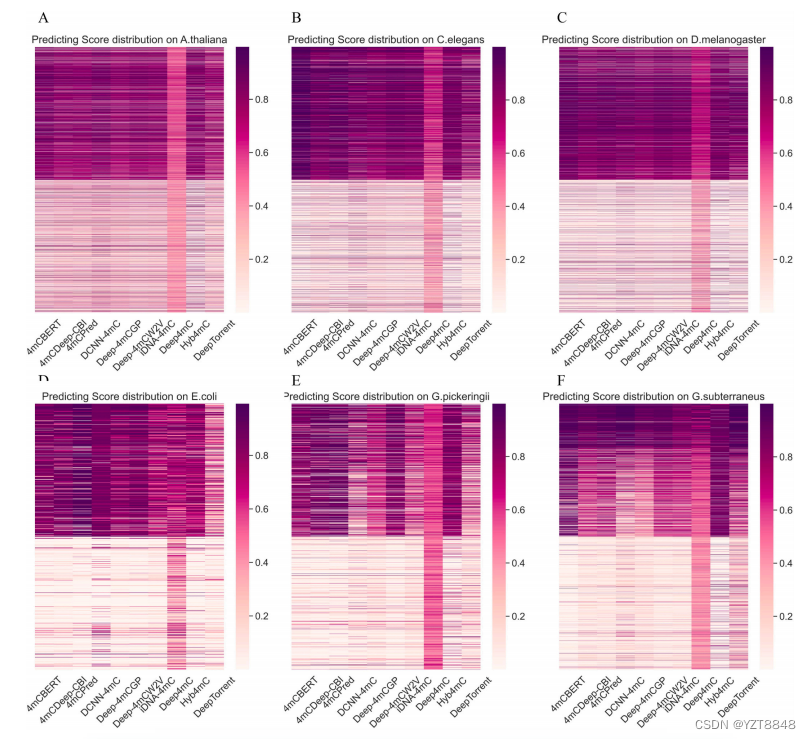
预测比较方法的得分分布(热图)
预测分数表明DNA序列段是否含有4mC位点的可能性。预测得分分布的热图直观地反映了每种方法对4mC站点的判别。如图9所示,颜色越深表示该DNA片段为4mC的概率越大,颜色越浅表示非4mC的概率越大。第一列的4mCBERT显示了多个物种上4mC和非4mC之间的明确界限。
不同截止阈值的性能
不同物种的最佳预测能力的截止值可能不同,不恰当的截止值将导致预测结果较差。
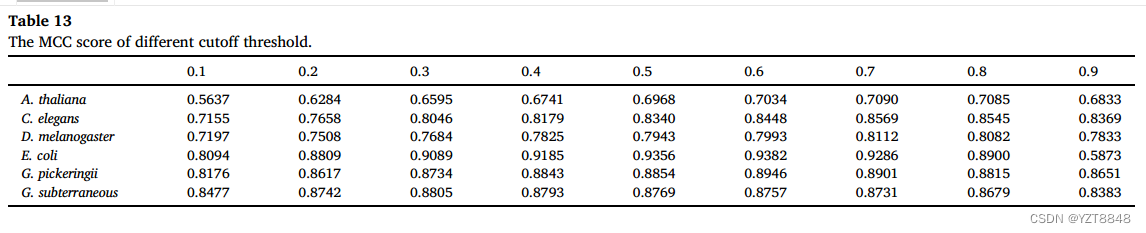
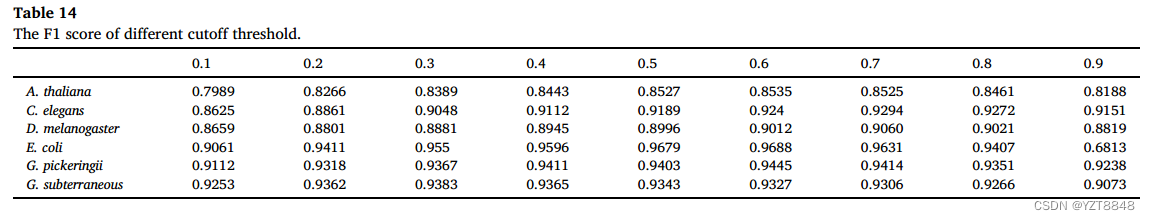
我们在表13和表14中给出了6个物种对应于不同截断值的MCC和F1分数。当MCC和F1达到最佳性能时,截断值范围为0.3 ~ 0.7。本文以0.5为临界值,4mC站点的预测得分大于等于0.5,非4mC站点的预测得分小于0.5。截止值的详细评估结果见表S18至S23。
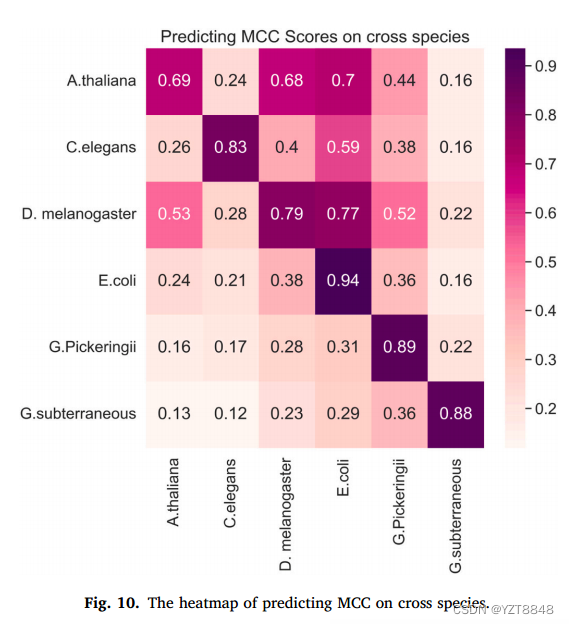
3.9. 跨物种实验验证了4mCBERT的泛化性。当一个模型在一个物种的数据集上训练,然后识别其他物种的4mC位点时,我们使用MCC来衡量鲁棒性。MCC的跨种热图如图10所示。可以观察到,4mCBERT在其自身物种上表现最好,而在除大肠杆菌物种外的其他物种数据集上表现平平。大肠杆菌模型在D. melanogaster上进行了测试,与D. melanogaster模型自我相比,得到了接近MCC的分数。此外,大肠杆菌模型在拟单胞菌数据集上进行了测试,甚至超过拟单胞菌模型本身。但是,C. elegans模型,G. pickeringii模型和G.。
地下模型对其他物种的鲁棒性较低,表明这些物种的4mC段模式与其他物种有很大差异,不适用于未标注的4mC段。此外,A. thaliana模型也没有表现出出色和稳健的性能。主要原因可能是种间同源性较低。总之,建议用他们自己训练的模型来预测一个4mC段。
跨物种数据集的详细评估结果见表S24至S29。
跨物种热图
跨物种综合数据集比较
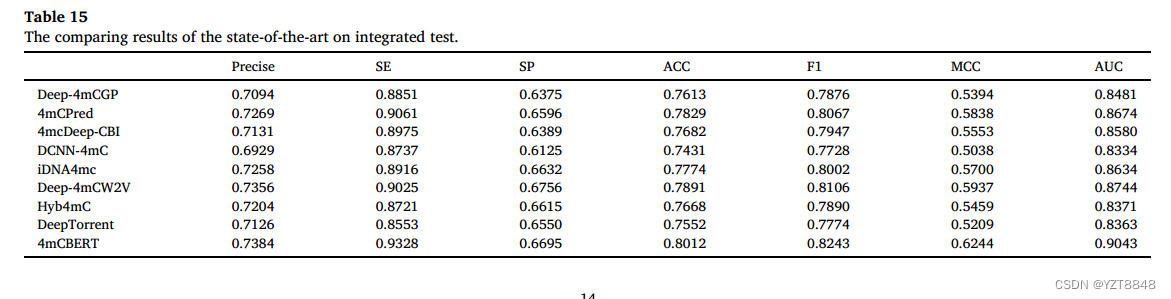
近年来,4mC位点的功能被发现并证明与许多生命过程有关。然而,在许多物种中,4mC位点没有很好地标注,这阻碍了进一步的研究。因此,我们整合了6个物种数据集,训练了一个未注释物种4mC位点预测的集成模型。首先,我们将6个物种训练数据集合并为一个综合训练数据集,并将6个物种测试数据集合并为一个综合测试数据集。其次,为了比较4mCBERT在综合独立测试数据集上的性能,引入Deep- 4mcgp、4mCPred、4mcDeep-CBI、DCNN-4mC、iDNA4mc、Deep- 4mCW2V、Hyb4mC和DeepTorrent作为竞争对手,综合训练和独立测试数据集与4mCBERT相同。最终,每种方法的比较结果如表15所示。从表15中可以看出,4mCBERT在集成独立测试数据集中表现出更高的性能,precision提高了0.28%至4.5%,SE提高了2.6%至5.91,ACC提高了1.21%至5.81%,F1提高了1.37%至5.15%,MCC提高了3.07%至12.06%,AUC提高了2.99%至7.09%。我们还绘制了AUROC和预测得分分布,以显示每种方法的详细差异,如图11所示。
讨论
DNA甲基化是一种生物过程,甲基被添加到DNA分子中。甲基化可以在不改变序列的情况下改变DNA片段的活性。当位于基因启动子中时,DNA甲基化通常起抑制基因转录的作用。
在哺乳动物中,DNA甲基化对正常发育至关重要,并与几个关键过程有关,包括基因组印迹、x染色体失活、转座因子抑制、衰老和致癌。4mC是DNA甲基化类型之一,在生命过程中起着重要作用。如何快速、准确地检测4mC位点是后续研究的主要上游工作,包括病理发现、癌症发病机制等。为了检测4mC位点,提取胞嘧啶上游20个核苷酸和下游20个核苷酸,生成41个核苷酸DNA片段。
因此,利用DNA片段提取的特征和机器学习模型,学习4mC和非4mC的不同模式,高通量检测4mC。在本研究中,我们建立了一个名为4mCBERT的集成学习模型来识别6个基准基因组数据集中的4mC位点。4mCBERT的工作流程可以概括为:1)比较常用的表示DNA序列片段的特征,过滤出有益的特征来识别4mC位点;2)首先构建了两类新的特征来识别4mC位点,包括PCP特征和化学BERT特征;3)与主流的机器和深度学习方法进行比较,我们发现集成学习模型获得了更好的性能;5)开发了4mCBERT的GUI软件和web服务器,便于4mC站点的预测和4mC识别模型的再训练。
虽然从DNA序列片段中可以提取出许多特征,但其中哪些特征有助于4mC位点的鉴定尚不清楚。因此,我们比较了6个基准数据集上14个广泛使用的特征。通过多指标比较,我们发现one-hot、EIIP、NCP和word2vec特征对识别4mC站点有积极贡献。此外,为了寻找新的贡献特征来识别4mC,我们根据核苷酸的化学性质提出了两种类型的特征。第一个提出的特征是PCP,它使用摩尔折射率、摩尔体积、等比体积、表面张力和极化率五种化学性质来表示一个信号核苷酸来编码一个DNA序列片段。第二个提出的特征是基于核苷酸的分子结构。“A”、“T”、“C”、“G”四种核苷酸可以用SMILES格式NC1=NC=NC2=C1N=CN2、O=C1C2NCNC2NC(N)N1、CC1=CNC(=O) NC1=O、NC1=C(NC(=O)N=C1)N表示,可视为一种特殊的生物信息学语言。采用BERT模型对各SMILES格式句子进行编码。与其他广泛应用的特征相比,本文提出的两类特征PCP和化学BERT增强了预测能力。基于多基准数据集上的比较特征,选择one-hot、EIIP、NCP、word2vec、PCP和三种BERT模型对DNA序列段进行编码。
6个基准数据集上的重要特征如图S1所示。
为了寻找合适的机器学习模型,我们比较了多个机器学习和深度学习模型,例如ResNet101、SVM、RF等在相同的输入信息下。对比结果表明,集成学习模型CatBoost表现出更好的性能和鲁棒性。因此,基于所选择的特征和CatBoost模型,我们构建了4mCBERT来识别4mC站点。我们还将4mCBERT与9种最先进的方法进行了比较,以确保优势和可靠性。与这些方法相比,4mCBERT在多个指标上表现出色。对于MCC的综合度量,4mCBERT在六个独立数据集上超过了次优模型4.32%,2.52%,2%,6.63%,8.59%和8.45%。
对于ACC、F1和AUC等其他指标,4mCBERT也表现出更高的改善。为了直观地观察预测结果的分布,分布式热图显示4mCBERT能够区分出边界明显的4mC站点和非4mC站点。此外,我们还在6个独立的数据集上进行了不同截止阈值的实验,发现在6个物种数据集上,大多数时候0.5是一个合适的阈值。此外,我们还评估了4mCBERT的跨种预测能力,我们发现4mCBERT表现出了共同的性能,其主要原因可能是种同源性较低。为了促进跨学科研究,我们开发了一个用户友好的软件来预测4mC站点,可以在https://github上访问。
可以在没有服务器资源限制的情况下跨平台运行。此外,4mCBERT软件提供GUI,通过点击鼠标重新训练其他物种的机器模型。我们还通过web服务器http://cczubio提供了一种简单的预测方法。
/ 4 mcbert。4mCBERT的应用将极大地促进4mC场地的跨学科研究。
综上所述,基于精心选择和构建的特征和集成学习策略,4mCBERT在多个基准数据集上显示出更高的4mC站点预测精度。但是,5mC和6mA也是DNA甲基化,因此,同时发现它们是当务之急。
在未来的工作中,我们将重点开发一种同时检测5mC、6mA和4mC的新模型或平台。
结论
在本文中,我们提出了一个集成学习模型,旨在通过DNA序列片段预测4mC位点。为了发现识别4mC站点的有益特征,我们比较了6个基准数据集上14个广泛使用的特征。我们发现one-hot、EIIP、NCP和word2vec特征对4mC位点的识别有积极的贡献。此外,我们还从核苷酸的化学性质提出了PCP和化学BERT特征。与其他广泛应用的特征相比,所提出的两类特征PCP和化学BERT获得了令人满意的性能。在独立基准测试数据集上的比较结果表明,4mCBERT在六个独立测试数据集上取得了显著的改进。4的MCC mcbert显著优于其他模型a芥,秀丽隐杆线虫,d .腹,大肠杆菌,g·皮克林和g .隐匿的独立数据集由4.32%提高到24.39%,2.52%,31.65%,2%,16.49%,6.63%,至35.15点,分别8.59%到61.85%和8.45%到34.45%。4mCBERT软件与图形用户界面可在https://github.com/abcair/4mCBERT和网络服务器在http://cczubio.top/4mCBERT预测4mC网站。总之,4mCBERT对识别4mC位点具有较高的置信度。one-hot、EIIP、NCP、word2vec、PCP和chemical BERT在识别4mC位点方面有积极贡献,这些位点可以扩展其他文件来编码DNA序列片段。在未来的工作中,我们将重点开发一种同时检测5mC、6ma和4mC的新模型或平台,以关联DNA甲基化研究。


